Ein 3D Atlas der Embryonalentwicklung
Computergestützte Methode ermöglicht neue Einblicke in die Aktivität von Genen während der frühen Organentwicklung
Die Forschungsgruppe von Alexander Meissner am Max-Planck-Institut für molekulare Genetik (MPIMG) hat in Zusammenarbeit mit dem Broad Institute und der Universität Marseille "virtuelle 3D-Embryonen" entwickelt, mit denen Wissenschaftler*innen die räumliche Verteilung der Genexpression während der Entwicklung untersuchen können. Ihre Arbeit, die in Nature Genetics veröffentlicht wurde, enthüllt Details darüber, wie sich Gewebe im frühen Mausembryo entwickeln.
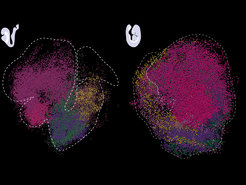
Unter dem Mikroskop erscheint die Welt der Zellen zweidimensional. In Wirklichkeit spielen sich biologische Prozesse in Geweben und Organen im dreidimensionalen Raum ab. Welche molekularen Mechanismen in einer einzelnen Zelle ablaufen, ist ebenso wichtig wie die Frage, wo sich eine Zelle innerhalb eines Gewebes oder Organs befindet und welche Gene zu einem bestimmten Zeitpunkt und an einem bestimmten Ort aktiv sind. Besonders wichtig ist dies in der frühen Entwicklung. Hier sind Zeitpunkt und Ort der Genaktivität entscheidend dafür, dass sich die verschiedenen Zelltypen bilden, aus denen später die Organe entstehen. "Embryonen wachsen sehr schnell", sagt Adriano Bolondi, Postdoc im Meissner-Labor und einer der Erstautoren. "Es ist daher wichtig, die Embryonalentwicklung aus einer multidimensionalen Perspektive zu betrachten, da sie von einer Vielzahl komplexer Inputs und Signalprozesse abhängt, die sonst übersehen werden könnten". Jüngste technologische Fortschritte haben es möglich gemacht, die Transkription von Genen in ihrer natürlichen Umgebung zu untersuchen - auch in sich entwickelndem Gewebe. Die Forschenden haben diesen Ansatz nun in die dritte Dimension gebracht.
Aus zweidimensionalen Schnappschüssen wird ein virtueller Embryo
Das Team nutzte Slide-seq, eine Methode, die von Fei Chen und Evan Macosko am Broad Institute entwickelt wurde. Das Prinzip hinter dieser Technologie ist einfach. Ausgangspunkt sind Gewebeproben von Mausembryonen, die jeweils einen "Schnappschuss" der Genaktivität in einem bestimmten Bereich enthalten. Mit Hilfe mikroskopisch kleiner Kügelchen, die mit DNA-Barcodes markiert sind, werden die Produkte der Gentranskription, die RNA-Moleküle, eingefangen und später sequenziert. Anhand dieser Daten können dann die Identität und die Menge der aktiven Gene an jedem Ort kartiert werden. "Einer der wichtigsten technologischen Fortschritte unserer Arbeit ist eine computergestützte Methode namens sc3D, die wir zusammen mit Léo Guignard, einem Computerbiologen der Universität Marseille, entwickelt haben. Damit können wir die zweidimensionalen Transkriptionsdaten in ein dreidimensionales Objekt umwandeln, das wir als "virtuellen Embryo" bezeichnen", erklärt Adriano Bolondi. Parallel zu diesem Ansatz entwickelte das Team eine Softwareanwendung, mit der die Expressionsmuster von rund 27.000 Genen im sich entwickelnden Embryo interaktiv erkundet werden können.
Einblicke in die Entwicklung des Nervensystems
Mit diesen Werkzeugen untersuchten die Wissenschaftler*innen die Entwicklung des Nervensystems im Detail. Dabei konzentrierten sie sich dabei auf das hintere Neuralrohr, eine Struktur, aus der später das Rückenmark und eine Vielzahl von Nervenzellen entstehen. Sie rekonstruierten ausgeprägte Genexpressionsmuster in diesem Gewebe und kartierten bisher unbeschriebene Gene, die nur in bestimmten Teilen des Neuralrohrs aktiv sind. Die Entdeckung dieser Gene wirft sofort Fragen nach ihrer genauen Funktion auf, denen die Wissenschaftler nun weiter nachgehen wollen. Schließlich charakterisierten sie mit ihrer Methode die Transkriptionslandschaft einer für den Embryo tödlichen Genmutation, bei der während der Embryogenese zusätzliche Neuralrohre entstehen. "Insgesamt erhöht unsere Methode die Granularität, mit der wir die Entwicklung von Säugetieren untersuchen, aber auch Defekte in der Embryogenese rekonstruieren können", sagt Adriano Bolondi. Die Forschenden hoffen nun, dass ihre Arbeit dazu beitragen kann, letztendlich eine vollständige Karte der Genregulation während der Entwicklung zu erstellen und Störungen und Entwicklungsdefekte während der Embryogenese zu erklären.












