3D-Struktur der DNA bildet umschriebenen Raum zur Aktivierung der Genexpression durch freie lncRNAs
Berliner Wissenschaftlerinnen und Wissenschaftler schlagen neue Form der Genregulation durch lange nicht-kodierende RNAs vor
Die DNA in unseren Zellen enthält alle Gene sowie lange Abschnitte, die nicht für Proteine kodieren, aber häufig regulatorische Funktionen im Genom haben. Eine Art von regulatorischen Abschnitten sind die Enhancer. Von ihnen werden sogenannte "lange nicht-codierende RNAs" (lncRNAs) abgeschrieben (transkribiert), die als entscheidende Regulatoren der Genexpression gelten. Forscherinnen und Forscher des Max-Planck-Instituts für molekulare Genetik (MPIMG) in Berlin konnten nun zeigen, dass eine lncRNA namens A-ROD nur funktionstüchtig ist, wenn sie vom Chromatin in das Kernplasma abgegeben wird. In der aktuellen Ausgabe der Zeitschrift Nature Communications beschreiben die Wissenschaftlerinnen und Wissenschaftler, dass sich A-ROD vollständig vom Chromatin ablösen muss, um regulatorisch aktiv werden zu können. Durch die dreidimensionale Struktur der DNA wird gewährleistet, dass sich A-ROD bei der Ablösung bereits in direkter Nähe zu seinem Zielgen befindet. Dies kann unser Verständnis der dynamischen Regulation der Genexpression in biologischen Prozessen stark beeinflussen.
Die Genome von Säugetieren enthalten nicht nur die Informationen für die Proteine des Organismus, sondern auch für Tausende von langen nicht-kodierenden RNAs (lncRNAs). Diese haben regulatorische Funktionen zum Beispiel bei der Entwicklung des Organismus oder der Entstehung von Krankheiten. lncRNAs entstehen mithilfe der gleichen molekularen Maschinerie wie die mRNAs. Um ihre Funktion auszuüben, verbleiben sie aber innerhalb des Zellkerns, wo sie mit Proteinen zusammenwirken und deren Bindung an die DNA vermitteln oder deren enzymatische Aktivität verstärken können. Sie sind häufig im Kern und am Chromatin angereichert, bislang war aber unklar, ob sie sich für ihre Funktion bei der Regulation der Transkription vom Chromatin ablösen müssen.
Jetzt haben die Forschungsgruppen „Lange nicht-kodierende RNAs“ unter der Leitung von Ulf Ørom (jetzt an der Universität Aarhus, Dänemark) und „RNA-Bioinformatik“ unter der Leitung von Annalisa Marsico eine lncRNA namens A-ROD (für Activating Regulator of DKK1) untersucht, die die Expression des DKK1-Gens verstärkt. Die Wissenschaftlerinnen und Wissenschaftler des Max-Planck-Instituts für molekulare Genetik (MPIMG) in Berlin konnten zeigen, dass A-ROD nur in dem Moment funktionsfähig ist, wenn es vom Chromatin in das Kernplasma abgegeben wird. Nur dann kann es Transkriptionsfaktoren - Proteine, die die Aktivität von Genen kontrollieren - an spezifische Stellen der DNA bringen, um so die Genexpression zu verstärken.
„Enhancer kontrollieren die Expression von Genen, die auf dem langgestreckten DNA-Faden weit von ihnen entfernt liegen“, erklärt Evgenia Ntini, Erstautorin der jetzt veröffentlichten Studie. „Interessanterweise stellten wir fest, dass lncRNAs von Enhancern, die mit ihren Zielgenen innerhalb einer gemeinsamen DNA-Schlaufe liegen, seltener an das Chromatin gebunden sind und sich innerhalb des Kernplasmas anreichern. Die Loslösung der lncRNA vom Chromatin scheint für ihre Funktion erforderlich zu sein.“
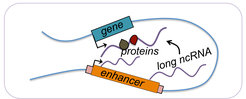
In der Chromatinform bildet der lineare DNA-Faden eine dreidimensionale Struktur mit festgelegten Schlaufen, wodurch genau definierte DNA-Abschnitte, die auf dem langgestreckten DNA-Faden weit voneinander entfernt liegen, in enge Nachbarschaft zueinander gelangen. Dies ist auch bei dem A-ROD-Enhancer und seinem Zielgen der Fall. „Innerhalb der Schlaufe kann A-ROD sofort mit dem DKK1-Gen und regulatorischen RNA-bindenden Proteinen zusammenwirken, um die Genexpression zu aktivieren“, sagt Ntini.
Auf der Grundlage ihrer Ergebnisse schlagen die Forscherinnen und Forscher eine neue Art der Genregulation vor, die durch lncRNAs vermittelt wird. Sie gehen davon aus, dass die lncRNA ihre Funktion nur ausüben kann, nachdem sie vollständig transkribiert ist und sich von der Transkriptionsstelle gelöst hat. Dabei muss sich der A-ROD-Enhancer in direkter Nähe zum DKK1-Gen befinden. Damit wäre nicht die Transkription der lncRNA der kritische Schritt für die Genaktivierung, sondern vielmehr ihre Freisetzung vom Chromatin, so dass sie für die Umgebung zugänglich wird, um regulatorische Proteine binden zu können.
Die Ergebnisse sind sowohl aus experimenteller als auch aus therapeutischer Sicht spannend, da die Ansätze zur gezielten Beeinflussung der RNA-Expression im Zytoplasma, im Kernplasma und im Chromatin sehr unterschiedlich sind. Die Forscherinnen und Forscher glauben, dass diese Unterschiede genutzt werden könnten, um die Ansätze zur gezielten Beeinflussung von RNA-abhängigen Prozessen bei Krankheiten zu optimieren. Als Nächstes wollen sie weitere Enhancer-ähnliche ncRNAs identifizieren, um ihr Potential und etwaige Einsatzmöglichkeiten für die Regulation der Genexpression zu untersuchen.











